细胞内由RNA聚合酶III(Pol III)参与的转录起始过程高度动态且复杂,包括转录起始蛋白因子对双链DNA的特异性识别,进而募集Pol III至转录起始位点。高等生物体内有一类含有III型启动子的基因,例如U6和7SK基因。Pol III转录起始这类基因不仅依赖普遍转录因子TFIIIB对TATA框的识别,同时还需要特异性转录因子小核RNA激活蛋白复合物(SNAPc)对目的基因5’端特异性元件(PSE)的识别。此类转录起始识别方式生成的非编码RNA产物与细胞生长调控密切相关。
尽管组成 SNAPc 复合物的各个亚基之间参与相互作用的结构域已被鉴定。SNAPC4作为核心组分直接参与和 SNAPC1、SNAPC3、SNAPC5 的相互作用。然而,SNAPc 复合物的组装模式、与启动子DNA的结合方式以及与TFIIIB和Pol III组装形成完整的转录起始转录起始复合物的方式仍然未知。我院徐彦辉团队过去两年来揭示了Pol III转录延伸(Cell Research,2021)和终止(Nature Communications,2021)复合物的结构和分子机制。
2023年5月10日,复旦大学生物医学研究院\复旦大学附属肿瘤医院徐彦辉团队在Cell Research杂志在线发表题为“Structure of the SNAPc-bound RNA polymerase III preinitiation complex”的研究论文。该研究利用冷冻电镜技术解析了SNAPc-TFIIIB-Pol III-U6的完整转录起始复合物结构,系统揭示了Pol III转录起始过程中启动子DNA的打开机制,以及SNAPc介导的转录起始分子机制。

该研究通过体外组装完整的转录起始复合物以及结构解析,捕获了七种不同构象的Pol III转录起始复合物。根据启动子DNA的构象,分为两种闭合状态(closed complex,CC),两种打开状态(open complex,OC)和三种打开中间状态(melting complex,OC)。结构分析表明,DNA的打开过程伴随着Pol III自身的一系列构象变化,包括在DNA 装载过程中,Pol III活性空腔的逐渐增大,由此以便于启动子DNA的高效结合;以及在DNA逐步打开直至形成稳定转录泡的过程中,Pol III活性空腔的逐减缩小以增加启动子DNA的结合稳定性。SNAPc复合物结合于U6启动子的PSE区域,由SNAPC3和SNAPC4的数个MYB结构域参与DNA的识别与结合。此外,SNAPc复合物与Pol III和TFIIIB直接相互作用以形成稳定的转录起始复合物。
此项工作首次解析获得了SNAPc依赖的Pol III转录起始复合物分子形貌,并捕获了DNA转录泡在转录起始阶段的形成过程,为全面解释U6 snRNA转录起始的分子机理提供的理论指导。
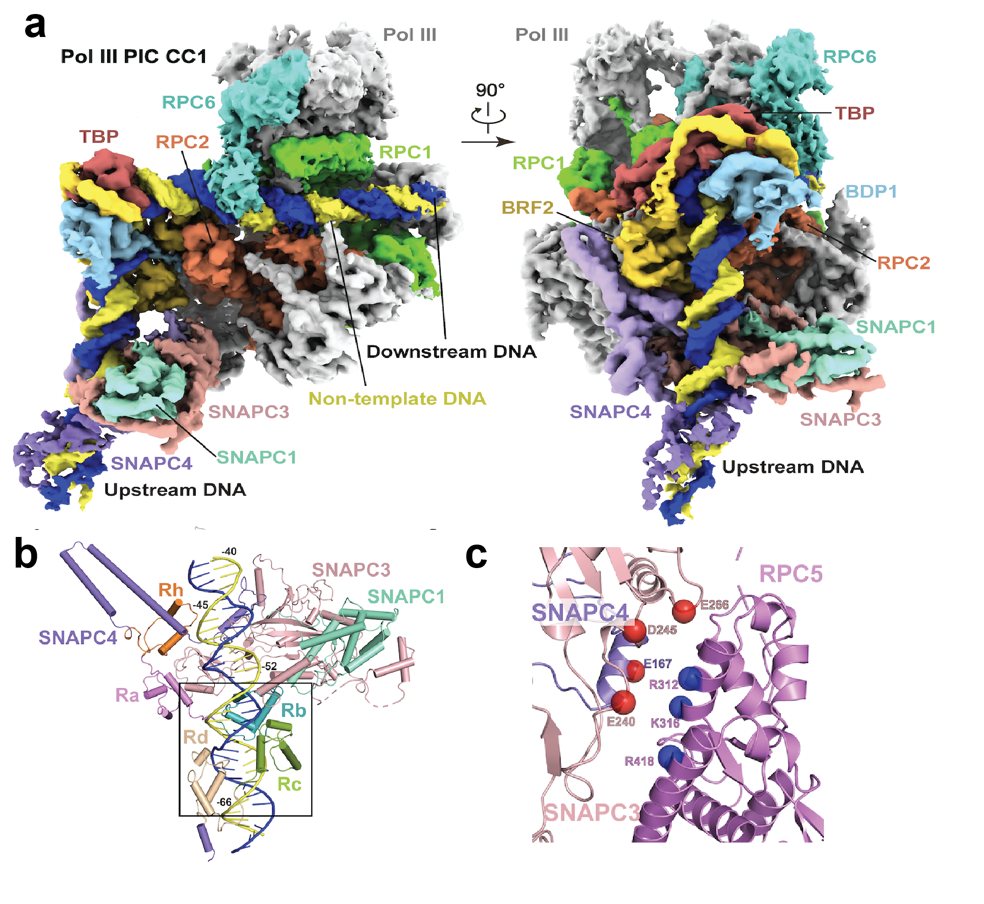
图1 a SNAPc-TFIIIB-Pol III-U6完整转录起始复合物的整体形貌;b SNAPc复合物结合U6启动子的三维模型;c SNAPc与Pol III的相互作用界面。
复旦大学附属肿瘤医院候海峰副研究员、生物医学研究院博士生金钱蔚和冷冻电镜平台任玉磊为本文共同第一作者。徐彦辉研究员和复旦大学生物医学研究院青年研究员王茜敏为本文的共同通讯作者。
论文链接:https://www.nature.com/articles/s41422-023-00819-x